[사전학습] 3.4 데이터 변환 - 특징 생성
3. 데이터 전처리 이해와 실무
3.1 데이터 변환 : 특징 생성
특징 생성 (Feature Creation)
- 원본 데이터의 조합/변환 등을 기반하여 새로운 특징들을 구축 및 생성하는 방법
=> 원본 데이터로 특징을 새롭게 생성하여 분석 과정 내 성능과 효율성 확보하고자 함목적 및 필요성
- 품질 확보 : 가공을 거치지 않은 Raw 데이터 활용 기반의 모델링은 품질 확보 어려움
- 최적화된 형태 변환 : 효과적인 Feature를 확보하는 것이 데이터 분석 내 가장 중요한 과정임
특징 생성 방안
- 범주 인코딩
- 크게 Nominal(순서가 없는)과 Ordinal(순서가 있는) 형식으로 나뉘는 범주형 변수
- 숫자가 아닌 범주 변수 값을 숫자로 표현하고 모델링에 적용하기 위한 과정
- 결합 및 분해
- 데이터 셋의 변수들의 조합을 기반으로 새로운 특징을 구축하는 방법
- 변수 간의 연산 혹은 분해를 통해 새로운 특징을 구축하고 입력 변수로 모델링에 적용
- 차원 축소
- 원본 데이터로부터 새로운 특징의 집합을 생성하는 것
- 고차원 원시 데이터 셋을 저차원으로 차원 축소하도록 새로운 특징을 생성하는 방식
1. 범주 인코딩
범주형 데이터의 알고리즘 적용을 위한 수치형 변환
One-hot Encoding
- 순서의 의미를 지니지 않은 범주형 변수를 처리하는 대표적 방법
- k개의 범주를 지닌 범주형 변수를 k개의 변수로 변환
2-1. 결합 기반 특징 생성
변수 간의 결합을 통해 새로운 의미를 지닌 특징을 생성
- Add / Divide / Subtract / Multiply
2-2. 분해 기반 특징 생성
변수의 분해를 통해 새로운 의미를 지닌 특징을 생성
- 특정 변수 활용 기반의 새로운 의미를 파악할 수 있는 특징을 생성하는 방법
- 도메인 지식 및 일반적 개념 기반으로 생성 가능 ex) 시간 -> 시간대, 요일 등..
3. 차원 축소 목적 특징 생성
3-1. 변수들이 지닌 정보를 최대한 확보하는 저차원 데이터로 생성
PCA(Principal Component Analysis)
- 서로 연관된 변수들이 관측되었을 때, 원본 데이터 분산 기반의 특징을 생성
- 주성분 간의 서로 독립을 이루도록 구성 (상관관계가 없도록 구성) 3-2. 군집 분석 기반의 고차원 데이터를 하나의 특징으로 차원 축소
Featurization via Clustering
- 고차원 데이터를 군집 분석을 기반으로 특징의 개수를 하나의 특징(군집 결과)으로 축소
- 이렇게 획득한 군집 결과 특징을 분류/회귀 등 문제 해결을 위한 입력 변수로 할용(Stacking 방법)
- 즉, 원본 데이터 내 여러 개의 특징을 하나의 특징으로 축소하여 모델 연산 비용 감소 추구
실습
샘플 데이터 필요
1
2
import numpy as np
import pandas as pd
1
2
data = pd.read_csv("./data/encoding_sample_data.csv", encoding='cp949')
data.head()
1
data['city'].value_counts()
1. OneHot Encoding
1
2
3
4
5
6
# city라는 범주형 변수 one_hot Encoding
# Pandas의 get_dummies 함수를 활용 쉽게 구혀 ㄴ가능
encoding_data = data.copy()
encoding_data = pd.get_dummies(encoding_data, columns=['city'])
encoding_data.head()
2. 결합 및 분해 기반 특징 생성
1
2
3
# 시간대 별 파악 목적
creation_data = data.copy()
creation_data.info()
1
2
3
# date 컬럼을 datetime 형식으로 변환
creation_data['date'] = pd.to_datetime(creation_data['date'])
creation_data.head()
1
creation_data.info()
1
2
3
4
5
6
creation_data['year'] = creation_data['date'].dt.year #연도
creation_data['month'] = creation_data['date'].dt.month #월
creation_data['day'] = creation_data['date'].dt.day #일
creation_data['hour'] = creation_data['date'].dt.hour #시간
creation_data['dayofweek'] = creation_data['date'].dt.dayofweek #요일 (월 = 0)
creation_data.head()
1
2
3
4
# AM PM
creation_data['ampm'] = 'AM'
creation_data.loc[creation_data['hour'] > 12, 'ampm'] = 'PM'
creation_data
3. 차원 축소 기반 특징 생성 (1) : PCA (주성분 분석)
- 여러 개의 변수를 지닌 고차원 데이터를 저차원으로 변환하도록 주성분들을 생성하는 알고리즘
- 원 변수들이 지닌 정보를 최대한 확보하는 저차원 데이터로 생성하는 방법
1
2
3
4
5
6
7
8
9
10
11
12
13
14
cancer = pd.read_csv('./data/wdbc.data', header=None)
# 데이터 컬럼명 지정
cancer.columns = [
"id", "diagnosis", "radius_mean", "texture_mean", "perimeter_mean", "area_mean", "smoothness_mean", "compactness_mean",
"concavity_mean", "concave_points_mean", "symmetry_mean", "fractal_dimension_mean", "radius_se", "texture_se",
"perimeter_se", "texture_worst", "smoothness_se", "compactness_se", "concavity_se", "concave_points_se", "symmetry_se",
"fractal_dimension_se", "radius_worst", "texture_worst", "perimeter_worst", "area_worst", "smoothness_worst", "compactness_worst",
"concavity_worst", "concave_points_worst", "symmetry_worst", "fractal_dimension_worst"
]
# ID를 Index화
cancer = cancer.set_index('id')
cancer
| diagnosis | radius_mean | texture_mean | perimeter_mean | area_mean | smoothness_mean | compactness_mean | concavity_mean | concave_points_mean | symmetry_mean | ... | radius_worst | texture_worst | perimeter_worst | area_worst | smoothness_worst | compactness_worst | concavity_worst | concave_points_worst | symmetry_worst | fractal_dimension_worst | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| id | |||||||||||||||||||||
| 842302 | M | 17.99 | 10.38 | 122.80 | 1001.0 | 0.11840 | 0.27760 | 0.30010 | 0.14710 | 0.2419 | ... | 25.380 | 17.33 | 184.60 | 2019.0 | 0.16220 | 0.66560 | 0.7119 | 0.2654 | 0.4601 | 0.11890 |
| 842517 | M | 20.57 | 17.77 | 132.90 | 1326.0 | 0.08474 | 0.07864 | 0.08690 | 0.07017 | 0.1812 | ... | 24.990 | 23.41 | 158.80 | 1956.0 | 0.12380 | 0.18660 | 0.2416 | 0.1860 | 0.2750 | 0.08902 |
| 84300903 | M | 19.69 | 21.25 | 130.00 | 1203.0 | 0.10960 | 0.15990 | 0.19740 | 0.12790 | 0.2069 | ... | 23.570 | 25.53 | 152.50 | 1709.0 | 0.14440 | 0.42450 | 0.4504 | 0.2430 | 0.3613 | 0.08758 |
| 84348301 | M | 11.42 | 20.38 | 77.58 | 386.1 | 0.14250 | 0.28390 | 0.24140 | 0.10520 | 0.2597 | ... | 14.910 | 26.50 | 98.87 | 567.7 | 0.20980 | 0.86630 | 0.6869 | 0.2575 | 0.6638 | 0.17300 |
| 84358402 | M | 20.29 | 14.34 | 135.10 | 1297.0 | 0.10030 | 0.13280 | 0.19800 | 0.10430 | 0.1809 | ... | 22.540 | 16.67 | 152.20 | 1575.0 | 0.13740 | 0.20500 | 0.4000 | 0.1625 | 0.2364 | 0.07678 |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 926424 | M | 21.56 | 22.39 | 142.00 | 1479.0 | 0.11100 | 0.11590 | 0.24390 | 0.13890 | 0.1726 | ... | 25.450 | 26.40 | 166.10 | 2027.0 | 0.14100 | 0.21130 | 0.4107 | 0.2216 | 0.2060 | 0.07115 |
| 926682 | M | 20.13 | 28.25 | 131.20 | 1261.0 | 0.09780 | 0.10340 | 0.14400 | 0.09791 | 0.1752 | ... | 23.690 | 38.25 | 155.00 | 1731.0 | 0.11660 | 0.19220 | 0.3215 | 0.1628 | 0.2572 | 0.06637 |
| 926954 | M | 16.60 | 28.08 | 108.30 | 858.1 | 0.08455 | 0.10230 | 0.09251 | 0.05302 | 0.1590 | ... | 18.980 | 34.12 | 126.70 | 1124.0 | 0.11390 | 0.30940 | 0.3403 | 0.1418 | 0.2218 | 0.07820 |
| 927241 | M | 20.60 | 29.33 | 140.10 | 1265.0 | 0.11780 | 0.27700 | 0.35140 | 0.15200 | 0.2397 | ... | 25.740 | 39.42 | 184.60 | 1821.0 | 0.16500 | 0.86810 | 0.9387 | 0.2650 | 0.4087 | 0.12400 |
| 92751 | B | 7.76 | 24.54 | 47.92 | 181.0 | 0.05263 | 0.04362 | 0.00000 | 0.00000 | 0.1587 | ... | 9.456 | 30.37 | 59.16 | 268.6 | 0.08996 | 0.06444 | 0.0000 | 0.0000 | 0.2871 | 0.07039 |
569 rows × 31 columns
1
2
3
4
5
6
# input 및 Target 구분
input_df = cancer.drop(['diagnosis'], axis=1)
print(np.shape(input_df))
target_df = cancer[['diagnosis']]
print(np.shape(target_df))
1
2
(569, 30)
(569, 1)
1
2
3
4
5
6
7
8
9
# 30개의 독립변수로 이루어진 데이터를 주성분 분석
# 주성분 분석 수행 이전, 각 변수의 스케일이 서로 다르기 때문에 표준화 수행
from sklearn.preprocessing import StandardScaler
std_scaler = StandardScaler()
std_scaler.fit(input_df)
input_scaled = std_scaler.transform(input_df)
input_scaled
1
2
3
4
5
6
7
8
9
10
11
12
13
array([[ 1.09706398, -2.07333501, 1.26993369, ..., 2.29607613,
2.75062224, 1.93701461],
[ 1.82982061, -0.35363241, 1.68595471, ..., 1.0870843 ,
-0.24388967, 0.28118999],
[ 1.57988811, 0.45618695, 1.56650313, ..., 1.95500035,
1.152255 , 0.20139121],
...,
[ 0.70228425, 2.0455738 , 0.67267578, ..., 0.41406869,
-1.10454895, -0.31840916],
[ 1.83834103, 2.33645719, 1.98252415, ..., 2.28998549,
1.91908301, 2.21963528],
[-1.80840125, 1.22179204, -1.81438851, ..., -1.74506282,
-0.04813821, -0.75120669]])
1
2
3
4
5
6
7
8
9
# 주성분 분석 수행
from sklearn.decomposition import PCA
# 두 개 주성분만 유지시키도록 수행
# 30개 변수의 데이터를 2개의 주성분으로 남도록 변환
pca = PCA(n_components=2)
pca.fit(input_scaled)
X_pca = pca.transform(input_scaled)
X_pca
1
2
3
4
5
6
7
array([[ 9.19283683, 1.94858307],
[ 2.3878018 , -3.76817174],
[ 5.73389628, -1.0751738 ],
...,
[ 1.25617928, -1.90229671],
[10.37479406, 1.67201011],
[-5.4752433 , -0.67063679]])
1
2
3
# PCA 수행된 input 데이터 확인
X_pca_df = pd.DataFrame(X_pca, columns=['pc1', 'pc2'])
X_pca_df
| pc1 | pc2 | |
|---|---|---|
| 0 | 9.192837 | 1.948583 |
| 1 | 2.387802 | -3.768172 |
| 2 | 5.733896 | -1.075174 |
| 3 | 7.122953 | 10.275589 |
| 4 | 3.935302 | -1.948072 |
| ... | ... | ... |
| 564 | 6.439315 | -3.576817 |
| 565 | 3.793382 | -3.584048 |
| 566 | 1.256179 | -1.902297 |
| 567 | 10.374794 | 1.672010 |
| 568 | -5.475243 | -0.670637 |
569 rows × 2 columns
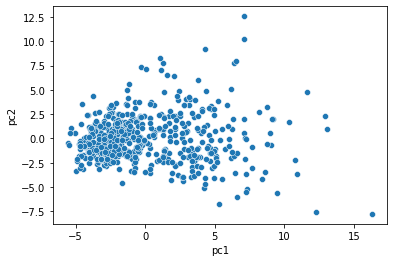
- 30개의 변수를 지닌 데이터가 2개의 특징들로 압축되었음
1
2
import matplotlib.pyplot as plt
import seaborn as sns
1
2
# 산점도로 2개의 주성분을 시각화
ax = sns.scatterplot(x='pc1', y='pc2', data=X_pca_df)
1
2
3
4
5
# Target과 확인을 위해 주성분 분석을 수행한 Input 데이터와 기존 Target 데이터를 Merge
target_df = target_df.reset_index()
pca_df = pd.merge(X_pca_df, target_df, left_index=True, right_index=True, how='inner')
pca_df = pca_df[['pc1', 'pc2', 'diagnosis']]
pca_df
| pc1 | pc2 | diagnosis | |
|---|---|---|---|
| 0 | 9.192837 | 1.948583 | M |
| 1 | 2.387802 | -3.768172 | M |
| 2 | 5.733896 | -1.075174 | M |
| 3 | 7.122953 | 10.275589 | M |
| 4 | 3.935302 | -1.948072 | M |
| ... | ... | ... | ... |
| 564 | 6.439315 | -3.576817 | M |
| 565 | 3.793382 | -3.584048 | M |
| 566 | 1.256179 | -1.902297 | M |
| 567 | 10.374794 | 1.672010 | M |
| 568 | -5.475243 | -0.670637 | B |
569 rows × 3 columns
1
2
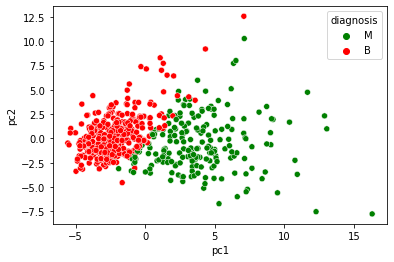
# 클래스를 색깔로 구분하여 처음 두개의 주성분으로 Target과 비교
ax = sns.scatterplot(x='pc1', y='pc2', hue='diagnosis', data=pca_df, palette=['green', 'red'])
- 실제 모델링에 적용하기 위해 효율적으로 활용 가능한 주성분 분석
특히, record 및 컬럼이 많은 경우 모델링 연산비용이 많이 들게 되므로 효율적인 차원 축소 기반의 특징을 생성하는 것이 분석 과정 내 필요한 과정
- 주성분을 선택하는 다른 방안
- 유지시킬 주성분 개수가 아닌 분산의 설명가능 수준을 비율로 선택 가능
- pca = PCA(n_components=0.8):
- 주성분의 수는 전체 분산의 최소 80% 수준에서 자동으로 선택
- 이를 통해 수치를 변경하면서 추출되는 주성분의 수 확인 가능하며, 이는 분산에 기초한 주성분 개수를 선택하는 부분에서 얼마나 많은 주성분을 사용할 것인지 확인해야 할 때 사용 가능
1
2
3
4
5
6
# 전체 분산의 최소 80% 수준에서 설명하는 수준의 주성분 확보
pca = PCA(n_components=0.8)
pca.fit(input_scaled)
X_pca = pca.transform(input_scaled)
X_pca_df = pd.DataFrame(X_pca)
X_pca_df
| 0 | 1 | 2 | 3 | 4 | |
|---|---|---|---|---|---|
| 0 | 9.192837 | 1.948583 | -1.123166 | 3.633731 | -1.195110 |
| 1 | 2.387802 | -3.768172 | -0.529293 | 1.118264 | 0.621775 |
| 2 | 5.733896 | -1.075174 | -0.551748 | 0.912083 | -0.177086 |
| 3 | 7.122953 | 10.275589 | -3.232790 | 0.152547 | -2.960878 |
| 4 | 3.935302 | -1.948072 | 1.389767 | 2.940639 | 0.546747 |
| ... | ... | ... | ... | ... | ... |
| 564 | 6.439315 | -3.576817 | 2.459487 | 1.177314 | -0.074824 |
| 565 | 3.793382 | -3.584048 | 2.088476 | -2.506028 | -0.510723 |
| 566 | 1.256179 | -1.902297 | 0.562731 | -2.089227 | 1.809991 |
| 567 | 10.374794 | 1.672010 | -1.877029 | -2.356031 | -0.033742 |
| 568 | -5.475243 | -0.670637 | 1.490443 | -2.299157 | -0.184703 |
569 rows × 5 columns
3. 차원 축소 기반 특징 생성 (2) : clustering (군집 분석)
- 여러 개의 변수를 하나의 변수(군집결과)로 변환 차원 축소
1
from sklearn.cluster import KMeans
1
2
3
# 일부 변수만 선택 (30개 변수 중 15개의 변수만 임의로 선정)
subset_df = input_df.iloc[:, 0:15]
subset_df
| radius_mean | texture_mean | perimeter_mean | area_mean | smoothness_mean | compactness_mean | concavity_mean | concave_points_mean | symmetry_mean | fractal_dimension_mean | radius_se | texture_se | perimeter_se | texture_worst | smoothness_se | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| id | |||||||||||||||
| 842302 | 17.99 | 10.38 | 122.80 | 1001.0 | 0.11840 | 0.27760 | 0.30010 | 0.14710 | 0.2419 | 0.07871 | 1.0950 | 0.9053 | 8.589 | 153.40 | 0.006399 |
| 842517 | 20.57 | 17.77 | 132.90 | 1326.0 | 0.08474 | 0.07864 | 0.08690 | 0.07017 | 0.1812 | 0.05667 | 0.5435 | 0.7339 | 3.398 | 74.08 | 0.005225 |
| 84300903 | 19.69 | 21.25 | 130.00 | 1203.0 | 0.10960 | 0.15990 | 0.19740 | 0.12790 | 0.2069 | 0.05999 | 0.7456 | 0.7869 | 4.585 | 94.03 | 0.006150 |
| 84348301 | 11.42 | 20.38 | 77.58 | 386.1 | 0.14250 | 0.28390 | 0.24140 | 0.10520 | 0.2597 | 0.09744 | 0.4956 | 1.1560 | 3.445 | 27.23 | 0.009110 |
| 84358402 | 20.29 | 14.34 | 135.10 | 1297.0 | 0.10030 | 0.13280 | 0.19800 | 0.10430 | 0.1809 | 0.05883 | 0.7572 | 0.7813 | 5.438 | 94.44 | 0.011490 |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 926424 | 21.56 | 22.39 | 142.00 | 1479.0 | 0.11100 | 0.11590 | 0.24390 | 0.13890 | 0.1726 | 0.05623 | 1.1760 | 1.2560 | 7.673 | 158.70 | 0.010300 |
| 926682 | 20.13 | 28.25 | 131.20 | 1261.0 | 0.09780 | 0.10340 | 0.14400 | 0.09791 | 0.1752 | 0.05533 | 0.7655 | 2.4630 | 5.203 | 99.04 | 0.005769 |
| 926954 | 16.60 | 28.08 | 108.30 | 858.1 | 0.08455 | 0.10230 | 0.09251 | 0.05302 | 0.1590 | 0.05648 | 0.4564 | 1.0750 | 3.425 | 48.55 | 0.005903 |
| 927241 | 20.60 | 29.33 | 140.10 | 1265.0 | 0.11780 | 0.27700 | 0.35140 | 0.15200 | 0.2397 | 0.07016 | 0.7260 | 1.5950 | 5.772 | 86.22 | 0.006522 |
| 92751 | 7.76 | 24.54 | 47.92 | 181.0 | 0.05263 | 0.04362 | 0.00000 | 0.00000 | 0.1587 | 0.05884 | 0.3857 | 1.4280 | 2.548 | 19.15 | 0.007189 |
569 rows × 15 columns
1
2
3
std_scaler.fit(subset_df)
subset_input_scaled = std_scaler.transform(subset_df)
subset_input_scaled
1
2
3
4
5
6
7
8
9
10
11
12
13
array([[ 1.09706398, -2.07333501, 1.26993369, ..., 2.83303087,
2.48757756, -0.21400165],
[ 1.82982061, -0.35363241, 1.68595471, ..., 0.26332697,
0.74240195, -0.60535085],
[ 1.57988811, 0.45618695, 1.56650313, ..., 0.8509283 ,
1.18133606, -0.29700501],
...,
[ 0.70228425, 2.0455738 , 0.67267578, ..., 0.27669279,
0.1806983 , -0.37934168],
[ 1.83834103, 2.33645719, 1.98252415, ..., 1.43852964,
1.0095027 , -0.17299998],
[-1.80840125, 1.22179204, -1.81438851, ..., -0.15744905,
-0.46615196, 0.04934236]])
1
2
3
4
# K-means 클러스터링 활용
# 군집 Label 수 설정
k = 5
model = KMeans(n_clusters=k, random_state=10)
1
2
3
model.fit(subset_input_scaled)
target_df['cluster'] = model.fit_predict(subset_input_scaled)
1
2
3
4
c:\Users\zxwlg\miniconda3\envs\dicom\lib\site-packages\sklearn\cluster\_kmeans.py:1334: UserWarning: KMeans is known to have a memory leak on Windows with MKL, when there are less chunks than available threads. You can avoid it by setting the environment variable OMP_NUM_THREADS=3.
warnings.warn(
c:\Users\zxwlg\miniconda3\envs\dicom\lib\site-packages\sklearn\cluster\_kmeans.py:1334: UserWarning: KMeans is known to have a memory leak on Windows with MKL, when there are less chunks than available threads. You can avoid it by setting the environment variable OMP_NUM_THREADS=3.
warnings.warn(
1
target_df
| index | id | diagnosis | cluster | |
|---|---|---|---|---|
| 0 | 0 | 842302 | M | 0 |
| 1 | 1 | 842517 | M | 2 |
| 2 | 2 | 84300903 | M | 2 |
| 3 | 3 | 84348301 | M | 4 |
| 4 | 4 | 84358402 | M | 2 |
| ... | ... | ... | ... | ... |
| 564 | 564 | 926424 | M | 0 |
| 565 | 565 | 926682 | M | 2 |
| 566 | 566 | 926954 | M | 2 |
| 567 | 567 | 927241 | M | 0 |
| 568 | 568 | 92751 | B | 3 |
569 rows × 4 columns
1
pd.crosstab(target_df.diagnosis, target_df.cluster)
| cluster | 0 | 1 | 2 | 3 | 4 |
|---|---|---|---|---|---|
| diagnosis | |||||
| B | 0 | 111 | 0 | 229 | 17 |
| M | 36 | 5 | 93 | 26 | 52 |
- 임의의 15개 변수만을 활용한 하나의 특징(군집 결과)이 Target 구분에 효과적일 것임을 예측 가능함
- 이처럼 많은 변수를 하나의 특징으로 구성하고, 입력 데이터의 차원을 줄인다면 모델 연산 비용 절감에 효과적
This post is licensed under CC BY 4.0 by the author.