[사전학습] 4.2 일변량 비시각화
4.2 일변량 시각화
일변량 시각화
하나의 변수로 구성된 데이터를 전체적인 관점으로 파악할 수 있는 데이터 탐색 유형
=> 단일 변수 데이터를 다양한 그래프로 시각화하여 전체적인 관점으로 살펴보는 것이 목적
시각화 특징
| 시각화 | 비시각화 |
|---|---|
| - 직관적 이해 추구 - 정보 전달 시간 단축 | - 효과적인 데이터 요약 - 빠른 데이터 성격 파악 |
범주형 시각화
1. 파이차트
- 비율파악을 용이하게 할 수 있음
- 시각적 전달 효과가 떨어지는 경우 : 범주 별 빈도가 유사한 경우, 범주 수가 많은 경우
2. 막대그래프
- 범주의 수가 많거나 범주 별 크기가 유사할 경우에 비교 수월
- 범주 별 비교는 막대그래프 기반의 파악이 비교적 수월함
연속형 시각화
1. 히스토그램
- 관측된 연속형 데이터 값들의 분포 파악 가능
- 구간 별 분포 상태를 쉽게 알아 볼 수 있는 그래프
- 구간 내 속하는 자료의 수가 많고 적음을 쉽게 파악
- 막대그래프와 유사한 형태를 보유
- 관측된 연속형 데이터 값들의 분포를 분석하여 연속성 있는 확률 밀도 함수를 추정
- 관측된 데이터로부터 변수가 가질 수 있는 모든 값의 확률(밀도)을 추정하는 것
- 히스토그램의 한계점을 극복하기 위해 고안된 방안
- 구간 경계가 연속적이지 않음
- 구간 크기 및 시작 위치에 따라 분포가 다르게 나타남
- 고차원 데이터에 대한 메모리 문제 발생 가능성 존재
3. 팍스플롯
- 연속형 데이터의 양상을 직관적으로 파악할 수 있는 방안으로 5가지 요약치를 기반으로 생성되며 다양한 정보를 손쉽게 표현
- 중앙값
- 1분위수
- 3분위수
- 최대값 (IQR Value)
- 최소값 (IQR Value)
- 데이터의 개략적인 흩어짐의 형태 파악 및 IQR 기반의 이상치 판단에 용이함
실습
1
2
3
4
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
1
2
3
4
5
6
import warnings
from sklearn.datasets import load_boston
with warnings.catch_warnings():
warnings.filterwarnings("ignore")
data = load_boston()
1
2
3
4
5
6
X = pd.DataFrame(data.data, columns=data.feature_names)
y = pd.DataFrame(data.target, columns=['MEDV'])
housing = pd.merge(X, y, left_index=True, right_index=True, how='inner')
housing_data = housing.copy()
범주형 일변량 시각화
1
housing_data.describe()
| CRIM | ZN | INDUS | CHAS | NOX | RM | AGE | DIS | RAD | TAX | PTRATIO | B | LSTAT | MEDV | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| count | 506.000000 | 506.000000 | 506.000000 | 506.000000 | 506.000000 | 506.000000 | 506.000000 | 506.000000 | 506.000000 | 506.000000 | 506.000000 | 506.000000 | 506.000000 | 506.000000 |
| mean | 3.613524 | 11.363636 | 11.136779 | 0.069170 | 0.554695 | 6.284634 | 68.574901 | 3.795043 | 9.549407 | 408.237154 | 18.455534 | 356.674032 | 12.653063 | 22.532806 |
| std | 8.601545 | 23.322453 | 6.860353 | 0.253994 | 0.115878 | 0.702617 | 28.148861 | 2.105710 | 8.707259 | 168.537116 | 2.164946 | 91.294864 | 7.141062 | 9.197104 |
| min | 0.006320 | 0.000000 | 0.460000 | 0.000000 | 0.385000 | 3.561000 | 2.900000 | 1.129600 | 1.000000 | 187.000000 | 12.600000 | 0.320000 | 1.730000 | 5.000000 |
| 25% | 0.082045 | 0.000000 | 5.190000 | 0.000000 | 0.449000 | 5.885500 | 45.025000 | 2.100175 | 4.000000 | 279.000000 | 17.400000 | 375.377500 | 6.950000 | 17.025000 |
| 50% | 0.256510 | 0.000000 | 9.690000 | 0.000000 | 0.538000 | 6.208500 | 77.500000 | 3.207450 | 5.000000 | 330.000000 | 19.050000 | 391.440000 | 11.360000 | 21.200000 |
| 75% | 3.677083 | 12.500000 | 18.100000 | 0.000000 | 0.624000 | 6.623500 | 94.075000 | 5.188425 | 24.000000 | 666.000000 | 20.200000 | 396.225000 | 16.955000 | 25.000000 |
| max | 88.976200 | 100.000000 | 27.740000 | 1.000000 | 0.871000 | 8.780000 | 100.000000 | 12.126500 | 24.000000 | 711.000000 | 22.000000 | 396.900000 | 37.970000 | 50.000000 |
1
2
3
4
bins = [0, 20, 40, 60, 80, 100]
names = ['G1', 'G2', 'G3', 'G4', 'G5']
housing_data['AGE_G5'] = pd.cut(housing_data['AGE'], bins, labels=names)
housing_data
| CRIM | ZN | INDUS | CHAS | NOX | RM | AGE | DIS | RAD | TAX | PTRATIO | B | LSTAT | MEDV | AGE_G5 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 0.00632 | 18.0 | 2.31 | 0.0 | 0.538 | 6.575 | 65.2 | 4.0900 | 1.0 | 296.0 | 15.3 | 396.90 | 4.98 | 24.0 | G4 |
| 1 | 0.02731 | 0.0 | 7.07 | 0.0 | 0.469 | 6.421 | 78.9 | 4.9671 | 2.0 | 242.0 | 17.8 | 396.90 | 9.14 | 21.6 | G4 |
| 2 | 0.02729 | 0.0 | 7.07 | 0.0 | 0.469 | 7.185 | 61.1 | 4.9671 | 2.0 | 242.0 | 17.8 | 392.83 | 4.03 | 34.7 | G4 |
| 3 | 0.03237 | 0.0 | 2.18 | 0.0 | 0.458 | 6.998 | 45.8 | 6.0622 | 3.0 | 222.0 | 18.7 | 394.63 | 2.94 | 33.4 | G3 |
| 4 | 0.06905 | 0.0 | 2.18 | 0.0 | 0.458 | 7.147 | 54.2 | 6.0622 | 3.0 | 222.0 | 18.7 | 396.90 | 5.33 | 36.2 | G3 |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 501 | 0.06263 | 0.0 | 11.93 | 0.0 | 0.573 | 6.593 | 69.1 | 2.4786 | 1.0 | 273.0 | 21.0 | 391.99 | 9.67 | 22.4 | G4 |
| 502 | 0.04527 | 0.0 | 11.93 | 0.0 | 0.573 | 6.120 | 76.7 | 2.2875 | 1.0 | 273.0 | 21.0 | 396.90 | 9.08 | 20.6 | G4 |
| 503 | 0.06076 | 0.0 | 11.93 | 0.0 | 0.573 | 6.976 | 91.0 | 2.1675 | 1.0 | 273.0 | 21.0 | 396.90 | 5.64 | 23.9 | G5 |
| 504 | 0.10959 | 0.0 | 11.93 | 0.0 | 0.573 | 6.794 | 89.3 | 2.3889 | 1.0 | 273.0 | 21.0 | 393.45 | 6.48 | 22.0 | G5 |
| 505 | 0.04741 | 0.0 | 11.93 | 0.0 | 0.573 | 6.030 | 80.8 | 2.5050 | 1.0 | 273.0 | 21.0 | 396.90 | 7.88 | 11.9 | G5 |
506 rows × 15 columns
1
2
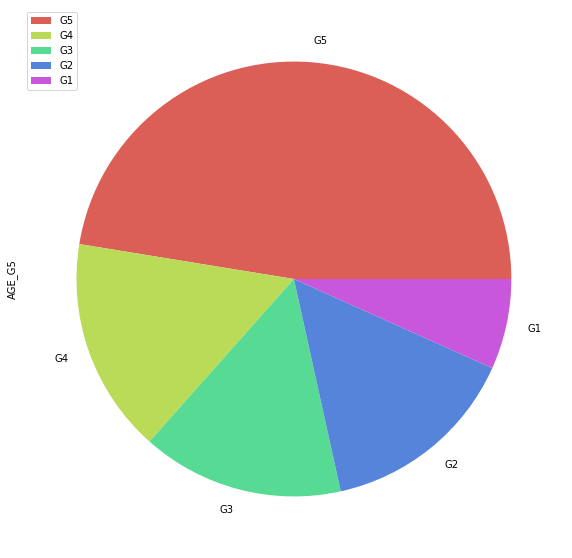
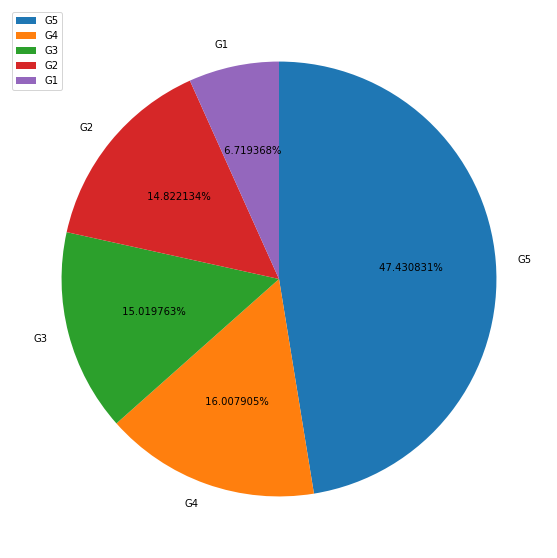
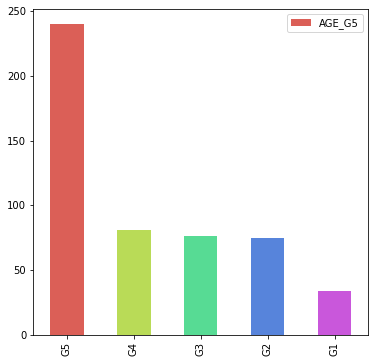
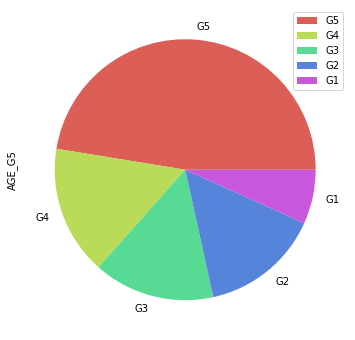
pie_df_G5 = pd.DataFrame(housing_data['AGE_G5'].value_counts())
pie_df_G5
| AGE_G5 | |
|---|---|
| G5 | 240 |
| G4 | 81 |
| G3 | 76 |
| G2 | 75 |
| G1 | 34 |
1
2
pie_df_G5.plot.pie(y='AGE_G5', figsize=(10, 10), colors=sns.color_palette('hls', len(pie_df_G5)))
plt.show()
1
2
3
4
plt.figure(figsize=(10, 10))
plt.pie(pie_df_G5['AGE_G5'], labels=pie_df_G5.index, startangle=90, counterclock=False, autopct='% 2f%%')
plt.legend()
plt.show()
1
2
3
4
5
6
7
8
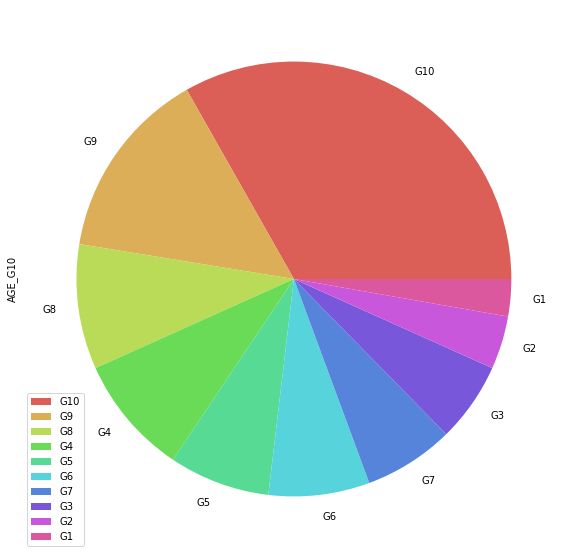
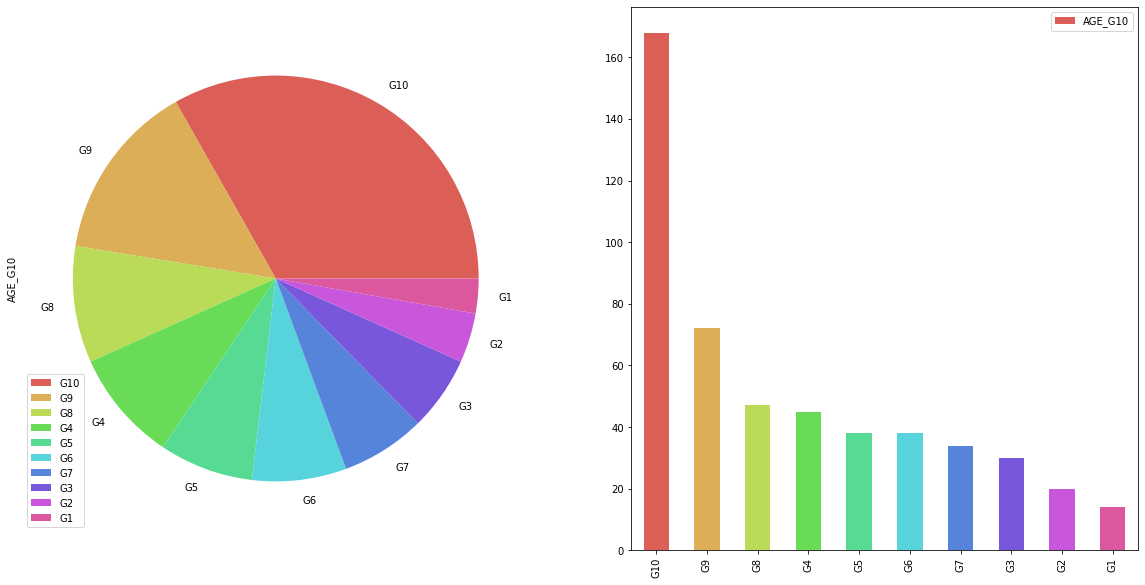
bins = list(np.linspace(0, 100, 11).astype(int))
names = ['G1', 'G2', 'G3', 'G4', 'G5', 'G6', 'G7', 'G8', 'G9', 'G10']
housing_data['AGE_G10'] = pd.cut(housing_data['AGE'], bins, labels=names)
pie_df_G10 = pd.DataFrame(housing_data['AGE_G10'].value_counts())
pie_df_G10.plot.pie(y='AGE_G10', figsize=(10, 10), colors=sns.color_palette('hls', len(pie_df_G10)))
plt.show()
- 5개의 범주인 경우보다, 범주 별 비교가 어려움을 보임
- 또한, G4~G7까지 대부분 비율이 유사하게 보이므로 비교가 쉽지 않음
- 데이터 크기가 유사할 경우, 막대그래프를 통해 크기 차이를 비교할 수 있음
막대그래프
1
2
pie_df_G5.plot.bar(y='AGE_G5', figsize=(6, 6), color=sns.color_palette('hls', len(pie_df_G5)))
plt.show()
1
2
pie_df_G5.plot.pie(y='AGE_G5', figsize=(6, 6), colors=sns.color_palette('hls', len(pie_df_G5)))
plt.show()
- G4, G3, G2의 비교가 어려운 파이차트 대비 막대그래프에서는 비교적 빈도 크기 비교가 수월함
1
2
3
4
5
fig, ax = plt.subplots(ncols=2, figsize=(20, 10))
pie_df_G10.plot.pie(y='AGE_G10', colors=sns.color_palette('hls', len(pie_df_G10)), ax=ax[0])
pie_df_G10.plot.bar(y='AGE_G10', color=sns.color_palette('hls', len(pie_df_G10)), ax=ax[1])
plt.show()
1
pie_df_G10
| AGE_G10 | |
|---|---|
| G10 | 168 |
| G9 | 72 |
| G8 | 47 |
| G4 | 45 |
| G5 | 38 |
| G6 | 38 |
| G7 | 34 |
| G3 | 30 |
| G2 | 20 |
| G1 | 14 |
연속형 일변량 시각화
1
2
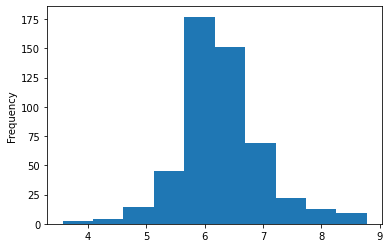
housing_data['RM'].plot.hist()
plt.show()
1
2
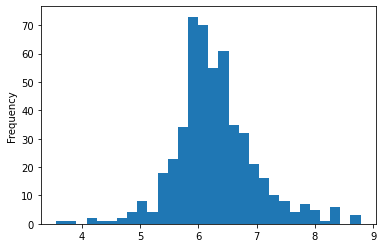
housing_data['RM'].plot.hist(bins=30)
plt.show()
커널밀도추정 시각화
1
2
3
4
5
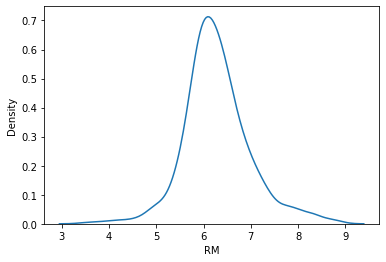
sns.kdeplot(housing_data['RM'])
plt.show()
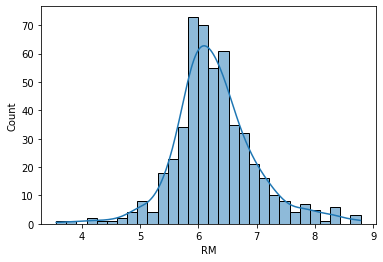
sns.histplot(housing_data['RM'], bins=30, kde=True)
plt.show()
- 관측된 RM 변수를 기반으로 커널밀도 추정 결과의 시각화
- 히스토그램 분포를 완만한 곡선으로 표현
1
2
3
4
5
6
7
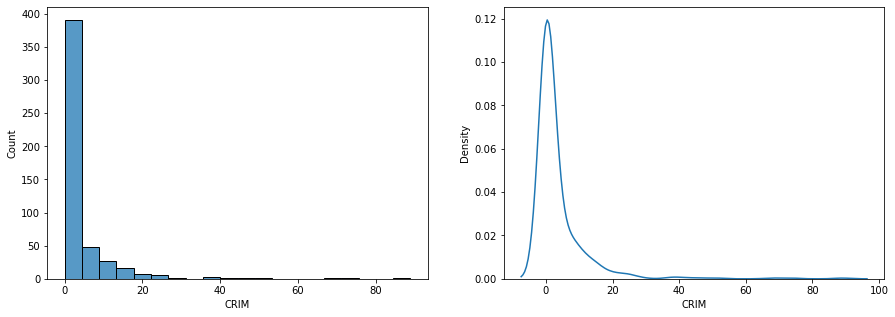
# CRIM 변수 시각화
fig, ax = plt.subplots(ncols=2, figsize=(15, 5))
sns.histplot(housing_data['CRIM'], bins=20, ax=ax[0])
sns.kdeplot(housing_data['CRIM'], ax=ax[1])
plt.show()
- CRIM 컬럼의 왜도 첨도 확인 결과, 왼쪽으로 치우쳐 있고 뾰족한 모양의 분포
- 시각화를 통해 실제 데이터 분포 형상 파악 수월
박스플롯
1
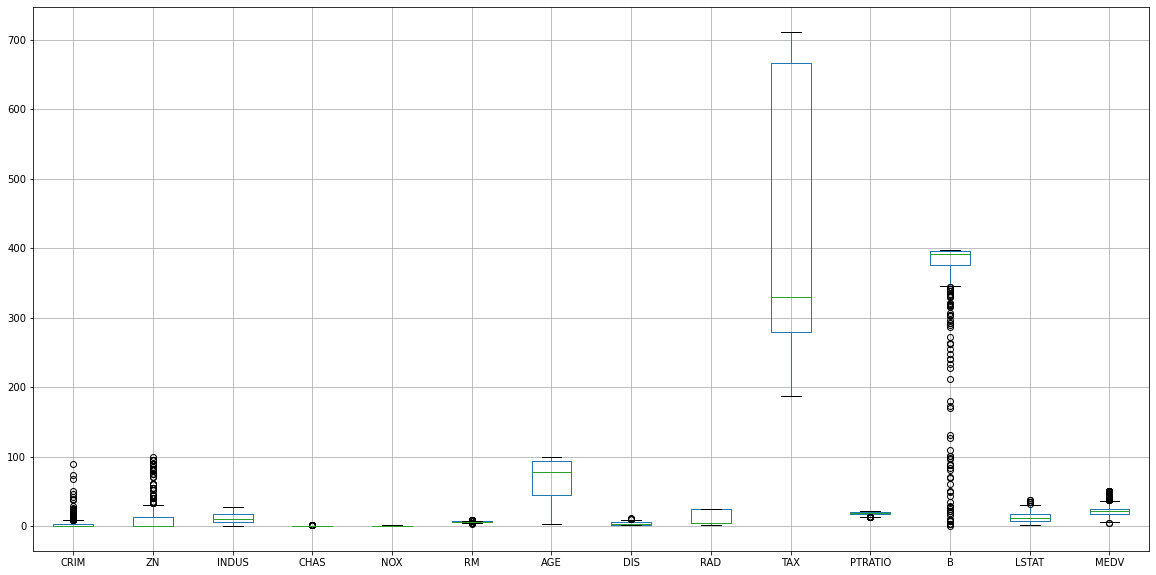
2
housing_data.boxplot(figsize=(20, 10))
plt.show()
- 변수 별 관측치 범위가 모두 다르기 때문에, 개략적 파악 가능
- 개별 변수 별 Box plot 시각화 필요
1
2
3
4
5
6
7
8
9
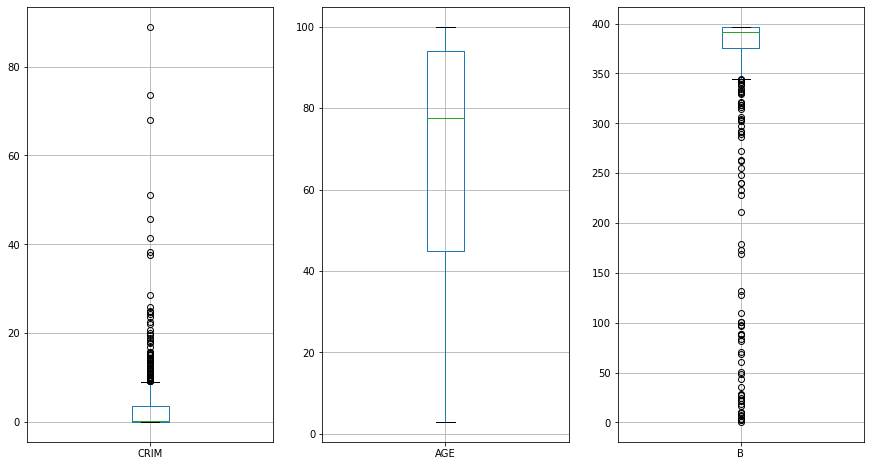
fig, ax = plt.subplots(ncols=3, figsize=(15, 8))
housing_data[['CRIM']].boxplot(ax=ax[0])
housing_data[['AGE']].boxplot(ax=ax[1])
housing_data[['B']].boxplot(ax=ax[2])
plt.show()
print(housing_data['AGE'].describe())
1
2
3
4
5
6
7
8
9
count 506.000000
mean 68.574901
std 28.148861
min 2.900000
25% 45.025000
50% 77.500000
75% 94.075000
max 100.000000
Name: AGE, dtype: float64
1
2
3
4
5
q1=housing_data['AGE'].quantile(0.25)
q3=housing_data['AGE'].quantile(0.75)
iqr=q3-q1
print('minimum :', q1-1.5*iqr)
print('maximum :', q3+1.5*iqr)
1
2
minimum : -28.54999999999999
maximum : 167.64999999999998
This post is licensed under CC BY 4.0 by the author.